
DNA
Sobre este escolas selecção Wikipedia
Crianças SOS, uma instituição de caridade educação , organizou esta selecção. SOS Children trabalha em 45 países africanos; você pode ajudar uma criança em África ?
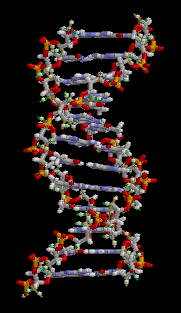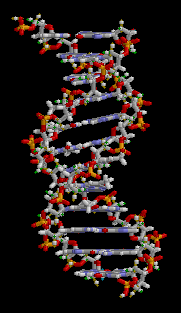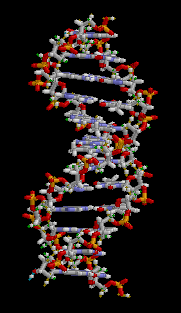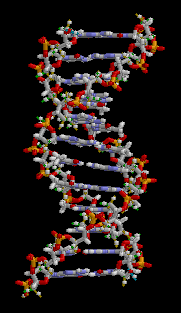
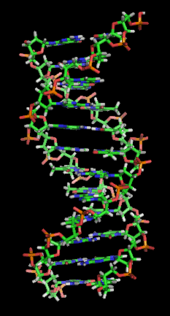
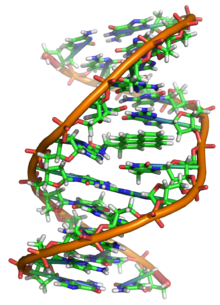
O ácido desoxirribonucléico (DNA) é uma molécula que codifica as genéticos instruções usadas no desenvolvimento e funcionamento de todos os vivos conhecidos organismos e muitos vírus . Juntamente com RNA e proteínas , o ADN é uma das três principais macromoléculas essenciais para todas as formas conhecidas de vida . A informação genética está codificada como uma sequência de nucleótidos ( guanina, adenina, timina, e citosina) registado utilizando as letras G, A, T e C. A maioria de ADN são moléculas de cadeia dupla hélice, que consiste em duas longo polímeros de unidades simples chamados nucleótidos, com moléculas backbones feitas de alternância açúcares ( desoxirribose) e grupos fosfato (relacionadas com ácido fosfórico), com a As nucleobases (G, A, T, C) ligados aos açúcares. ADN é bem adequada para armazenamento de informação biológica, uma vez que a espinha dorsal do ADN é resistente à clivagem e a estrutura de cadeia dupla fornece a molécula com um embutido duplicado da informação codificada.
Estas duas cadeias correm em direcções opostas umas às outras e são, por conseguinte, anti-paralelo, uma espinha dorsal de ser 3 '(três nobre) e os outros 5' (cinco nobre). Isto refere-se a direção é de frente para o carbono 3 e 5 na molécula de açúcar. Junto de cada açúcar é um dos quatro tipos de moléculas chamadas nucleobases (informal, bases). É o sequência destes quatro nucleobases ao longo da espinha dorsal que codifica a informação. Esta informação é lida usando o código genético , que especifica a sequência de aminoácidos dentro das proteínas. O código é lido copiando trechos de ADN no relacionada ARN de ácido nucleico em um processo chamado transcrição.
Dentro das células, o DNA é organizada em estruturas longas chamadas cromossomas. Durante divisão celular desses cromossomos são duplicados no processo de Replicação do ADN, fornecendo cada pilha seu próprio conjunto completo de cromossomos. organismos eucarióticos ( animais , plantas , fungos , e protistas) loja a maior parte de seu DNA dentro do núcleo da célula e algum do seu DNA em organelos, tais como mitocôndrias ou cloroplastos. Em contraste, procariontes ( bactérias e archaea) armazenam apenas o seu DNA no citoplasma. Dentro dos cromossomas, proteínas, tais como cromatina histones comprimem e organizam o ADN. Estas estruturas compactas guiam as interações entre o ADN e as outras proteínas, ajudando a controlar quais as partes de ADN são transcritas.
Propriedades

ADN é um longo polímero feito a partir de unidades de repetição chamados nucleótidos. O DNA foi identificado pela primeira vez e isoladas por Friedrich Miescher e a estrutura em dupla hélice do DNA foi descoberto pela primeira vez por James D. Watson e Francis Crick . A estrutura de ADN de todas as espécies compreende duas cadeias helicoidais cada uma enrolada em torno do mesmo eixo, e cada uma com um passo de 34 angstroms (3,4 nanómetros) e um raio de 10 angstroms (1,0 nanômetros). De acordo com outro estudo, quando medida numa solução em particular, a cadeia de ADN medido 22 a 26 angstroms de largura (2,2 a 2,6 nanômetros), e uma unidade de nucleótidos medido 3,3 Å (0,33 nm) de comprimento. Embora cada unidade de repetição individuais é muito pequena, polímeros de ADN podem ser moléculas muito grandes que contêm milhões de nucleótidos. Por exemplo, a maior humano cromossoma, cromossoma número 1, é de aproximadamente 220 milhões pares de bases de comprimento.
Em organismos vivos ADN não existir como uma única molécula, mas sim como um par de moléculas que são mantidos firmemente juntos. Estas duas longas cadeias entrelaçam como videiras, na forma de um dupla hélice. As repetições de nucleotídeos conter tanto o segmento da espinha dorsal da molécula, o que mantém a corrente em conjunto, e uma nucleobase, que interage com a outra cadeia de ADN em hélice. Um nucleobases ligada a um açúcar é chamado um nucleosídeo e uma base ligada a um açúcar e um ou mais grupos fosfato é chamado um nucleótidos. Um polímero compreendendo vários nucleótidos ligados (como em ADN) é chamado um polinucleótido.
A espinha dorsal da cadeia de ADN é feita a partir alternada fosfato e açúcar resíduos. O açúcar no ADN é 2-desoxirribose, que é um pentose (cinco carbono açúcar). Os açúcares são unidas entre si por grupos de fosfatos que formam ligações fosfodiéster entre os terceiro e quinto carbono átomos de anéis de açúcar adjacentes. Estes assimétrica títulos significar uma cadeia de ADN tem uma direção. Numa dupla hélice, a direcção dos nucleótidos em uma cadeia é oposta à sua direcção na outra cadeia: os fios são antiparalelas. As extremidades assimétricos de cadeias de ADN são chamados a 5 '(cinco prime) e 3 '(três privilegiada) termina, com a extremidade 5' com um grupo fosfato terminal e a extremidade 3 'de um grupo hidroxilo terminal. Uma diferença importante entre o ADN e o ARN é o açúcar, com a 2-desoxirribose no ADN a ser substituída pelo açúcar pentose alternativa ribose no ARN.
A dupla hélice do ADN é estabilizada principalmente por duas forças: pontes de hidrogênio entre nucleotídeos e interacções de empilhamento de bases entre nucleobases aromáticos. No ambiente aquoso da célula, o conjugado π ligações de bases de nucleótidos alinhar perpendicular ao eixo da molécula de ADN, minimizando a sua interacção com o camada de solvatação e, portanto, a energia livre de Gibbs . As quatro bases de ADN são encontrados adenina (abreviado A), citosina (C), guanina (G) e timina (T). Estes quatro bases estão ligados ao açúcar / fosfato para formar as sequências de nucleótidos completa, como mostrado, por monofosfato de adenosina.
Classificação de nucleobases
As nucleobases estão classificados em dois tipos: o purinas, A e G, sendo fundido com cinco e seis membros compostos heterocíclicos, e o pirimidinas, os anéis de seis membros de C e T. Um quinto de nucleobases pirimidina, uracilo (U), normalmente toma o lugar da timina em ARN e difere da timina por uma falta grupo metilo no seu anel. Além de ARN e ADN de um grande número de artificial análogos de ácidos nucleicos também foram criados para estudar as propriedades dos ácidos nucleicos, ou para utilização em biotecnologia.
Uracila normalmente não está presente no ADN, só ocorrendo como um produto da decomposição da citosina. No entanto numa série de bacteriófagos - Bacillus subtilis e bacteriófagos PBS1 PBS2 e Yersinia piR1-37 bacteriófago - timina foi substituída por uracilo. Uma forma modificada (beta-d-glucopyranosyloxymethyluracil) é também encontrado em vários organismos: os flagelados Diplonema e Euglena, e todo o cinetoplastida géneros Biossíntese de J ocorre em duas etapas: na primeira etapa de uma timidina no ADN específico é convertido em hydroxymethyldeoxyuridine; no segundo HOMedU é glicosilada para formar J. As proteínas que se ligam especificamente a esta base, foram identificados. Estas proteínas parecem ser parentes distantes do oncogene Tet1 que está envolvida na patogénese de leucemia mielóide aguda. J parece actuar como um sinal de terminação para ARN polimerase II.

Estrias
Fitas helicoidais gêmeas formam a espinha dorsal do DNA. Outra dupla hélice pode ser encontrada traçando os espaços ou ranhuras, entre os fios. Estas cavidades são adjacentes aos pares de bases e podem proporcionar um local de ligação. Como os fios não estão simetricamente localizados em relação um ao outro, as ranhuras são dimensionadas de forma desigual. Um sulco, o sulco maior, é de 22 Å de largura eo outro, o sulco menor, é de 12 Å de largura. A estreiteza do sulco menor, significa que as bordas das bases são mais acessíveis no sulco maior. Como resultado, as proteínas gosto factores de transcrição que se podem ligar a sequências específicas de ADN de cadeia dupla geralmente fazer contato com os sítios das bases expostos no sulco maior. Esta situação varia em conformações incomuns de ADN no interior da célula (ver abaixo), mas os sulcos maiores e menores são sempre nomeados para reflectir as diferenças em tamanho que seriam vistos se o DNA é torcido de volta para a forma B comum.
Base emparelhamento
Em uma hélice dupla do DNA, cada tipo de nucleobases em títulos uma cadeia com apenas um tipo de nucleobases na outra cadeia. Isto é chamado complementar emparelhamento base. Aqui, purinas formam pontes de hidrogênio com pirimidinas, com adenina ligação apenas para timina em duas pontes de hidrogênio, e citosina guanina ligação só para em três ligações de hidrogênio. Este arranjo de dois nucleotídeos complementares na dupla hélice é chamado par de base. Como ligações de hidrogênio não são covalente, podem ser quebradas e reunidas de forma relativamente fácil. As duas cadeias de ADN em dupla hélice pode ser separadas como um fecho de correr, ou por uma força mecânica ou alta temperatura . Como resultado desta complementariedade, toda a informação na sequência de cadeia dupla de uma hélice de DNA é duplicado em cada mecha, que é fundamental para a replicação do DNA. Com efeito, esta interacção específica e reversível entre pares de bases complementares é crítica para todas as funções do ADN em organismos vivos.
 |
 |
Os dois tipos de pares de base formam diferentes números de pontes de hidrogênio, AT forma duas pontes de hidrogênio enquanto que GC formam três pontes de hidrogênio (ver figuras, direita). ADN com alta GC-conteúdo é mais estável do que o DNA com baixo GC-conteúdo.
Como observado acima, a maioria das moléculas de ADN são, na verdade, dois cordões de polímero, ligados em conjunto de forma helicoidal através de ligações não covalentes; esta estrutura de cadeia dupla (dsDNA) é mantida em grande parte, pela base intracadeia empilhamento interações, que são mais forte para G, C pilhas. As duas fitas pode vir distante - um processo conhecido como fusão - para formar duas moléculas de ADNcs. Fusão ocorre quando as condições favorecem ssDNA; tais condições são temperatura elevada, baixo teor de sal e pH elevado (pH baixo também derrete ADN, mas uma vez que o ADN é instável devido a despurinação do ácido, pH baixo é raramente utilizado).
A estabilidade da forma dsDNA depende não só no conteúdo GC (% G, os pares de bases C), mas também em sequência (uma vez que o empilhamento é uma sequência específica) e também o comprimento (moléculas mais longas são mais estáveis). A estabilidade pode ser medida de várias maneiras; uma forma comum é a "temperatura de fusão", que é a temperatura à qual 50% das moléculas de ds são convertidos em moléculas ss; temperatura de fusão depende da força iónica e a concentração de ADN. Como resultado, é tanto a percentagem de pares de bases GC e o comprimento total de uma hélice dupla de ADN que determina a força de associação entre as duas cadeias de ADN. Hélices longas de DNA com um elevado teor de GC-fios mais fortes têm-interagindo, enquanto hélices curtas com alto teor de AT têm fios mais fracos interagindo. Em biologia, partes da dupla hélice do DNA que precisam separar facilmente, como o TATAAT Caixa Pribnow em alguns promotores, tendem a ter um elevado conteúdo de AT, fazendo com que os fios mais fáceis de separar.
No laboratório, a força desta interacção pode ser medida encontrando a temperatura necessária para quebrar as ligações de hidrogénio, os seus temperatura de fusão (também chamado valor de Tm). Quando todos os pares de bases de um ADN de dupla hélice, separar as cadeias e existem em solução como duas moléculas inteiramente independentes. Estes moléculas de ADN de cadeia simples (ssDNA) não têm uma única forma comum, mas algumas conformações são mais estáveis do que os outros.
Sentido e anti-
Uma sequência de ADN que é chamado de "sentido", se a sua sequência é a mesma que a de um RNA mensageiro cópia que é traduzido em proteína. A sequência da cadeia oposta é chamado a sequência "anti-sentido". Ambas as sequências com sentido e anti-sentido podem existir em diferentes partes da mesma cadeia de ADN (isto é, ambos os filamentos contêm ambas as sequências com sentido e anti-sentido). Em ambos os procariotas e eucariotas, as sequências de ARN anti-sentido são produzidos, mas as funções destes RNAs não são totalmente claras. Uma proposta é que o ARN anti-sentido estão envolvidos na regulação a expressão do gene através da base RNA-RNA emparelhamento.
A sequências de ADN em procariotas e eucariotas poucos, e mais em plasmídeos e vírus , borrar a distinção entre sentido e anti-fios por ter genes que se sobrepõem. Nestes casos, algumas sequências de ADN uma dupla função, que codifica uma proteína, quando lidas em conjunto uma cadeia, e uma segunda proteína quando lidas na direcção oposta ao longo da outra cadeia. Em bactérias , esta sobreposição pode estar envolvido na regulação da transcrição de genes, enquanto que no vírus, genes sobrepostos aumentar a quantidade de informação que pode ser codificados dentro do genoma viral pequena.
Supercoiling
O ADN pode ser torcido, como uma corda num processo denominado Superenrolamento do ADN. Com o ADN no seu estado "relaxada", uma fita normalmente circunda o eixo da hélice dupla, uma vez a cada 10,4 pares de bases, mas se o DNA está torcido as cadeias ficam mais ou menos enroladas. Se o DNA está torcido na direcção da hélice, isto é superenrolamento positivo, e as bases são unidas mais firmemente. Se eles são torcidos na direcção oposta, isto é o superenrolamento negativo, e as bases de se separar mais facilmente. Na natureza, o DNA apresenta um ligeiro supercoiling negativo que é causado por enzimas chamadas topoisomerases. Estas enzimas também são necessárias para aliviar o estresse de torção causado no DNA durante processos como transcrição e A replicação do ADN.

Estruturas de DNA suplentes
ADN existe em muitas possíveis conformações que incluam Uma ADN-, B-DNA, e Formas Z-DNA, apesar de apenas B-ADN e ADN-Z foram observados a partir de organismos funcionais. A conformação que adopta ADN depende do nível de hidratação, sequência de ADN, a quantidade e direcção de super-enrolamento, modificações químicas das bases, do tipo e da concentração de metal de iões , bem como a presença de poliaminas em solução.
Os relatórios publicados primeira de A-DNA Raios-X patterns- difração e também B-DNA - usado análises baseadas em Patterson transforma aquela fornecida apenas uma quantidade limitada de informações estruturais para fibras orientadas de DNA. Uma análise alternativo foi em seguida proposto por Wilkins et al., Em 1953, para o desenvolvimento in vivo de ADN-B de difracção de raios-X / espalhamento padrões de fibras de ADN altamente hidratadas em termos dos quadrados das funções de Bessel . Na mesma revista, James D. Watson e Francis Crick apresentou o seu análise de modelação molecular do DNA padrões de difracção de raios X que sugerem que a estrutura era uma hélice dupla.
Embora a forma `B-ADN" representa a mais comum nas condições encontradas nas células, não é uma conformação bem definida, mas uma família de conformações de ADN relacionadas que ocorrem nos níveis elevados de hidratação presentes em células vivas. Os correspondentes de difracção de raios X e dispersão padrões são característicos do moleculares paracrystals com um grau significativo de doença.
Em comparação com B-DNA, o formulário A-DNA é uma espiral dextra mais larga, com uma fenda menor larga e superficial e uma fenda maior estreita mais profundo. O Uma forma ocorre sob condições não fisiológicas em amostras parcialmente desidratados de ADN, enquanto que na célula pode ser produzida em emparelhamentos híbridos de ADN e ARN fios, bem como em complexos DNA-enzima. Segmentos de ADN, onde as bases foram quimicamente modificados pela metilação pode sofrer uma grande modificação na sua formação e adoptar a Forma Z. Aqui, os fios de rodar em torno do eixo helicoidal numa canhoto espiral, o oposto da forma mais comum B. Estas estruturas invulgares podem ser reconhecidas por proteínas de ligação de ADN-Z e podem estar envolvidas na regulação de transcrição.
Química DNA alternativo
Para um número de anos exobiologists propuseram a existência de um biosfera sombra, uma biosfera microbiana postulado da Terra que usa radicalmente diferentes processos bioquímicos e moleculares do que a vida atualmente conhecida. Uma das propostas foi a existência de formas de vida que utilizam arsénio em vez de fósforo no DNA. Um relatório em 2010 sobre a possibilidade de a bactéria GFAJ-1, foi anunciado, embora a pesquisa foi contestada, e as evidências sugerem que a bactéria evita ativamente a incorporação de arsénio na espinha dorsal do DNA e outras biomoléculas.
Estruturas Quadruplex
Nas extremidades dos cromossomas lineares são regiões especializadas de ADN chamados telômeros. A função principal destas regiões é para permitir que a célula para replicar extremidades do cromossoma usando a enzima telomerase, como as enzimas que permitem replicar DNA normalmente não pode copiar o extremo 3 'de cromossomos. Estas tampas de cromossoma especializadas também ajudam a proteger as extremidades do DNA, e parar os de reparo de DNA sistemas da célula as trate como danos a ser corrigido. Em células humanas, os telómeros são geralmente comprimentos de ADN de cadeia simples contendo vários milhares de repetições de uma sequência simples TTAGGG.
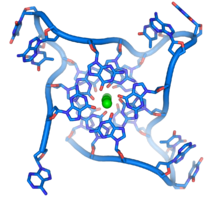
Estas sequências ricas em guanina podem estabilizar extremidades do cromossoma através da formação de estruturas de conjuntos empilhados de unidades de quatro bases, em vez de os pares de bases usuais encontrados em outras moléculas de DNA. Aqui, quatro bases de guanina formar uma chapa plana e estas unidades de quatro bases planas, em seguida, empilhados no topo uns dos outros, para formar um estábulo Estrutura G-quadruplex. Estas estruturas são estabilizadas por pontes de hidrogénio entre as bordas das bases e a quelação de um ião metálico no centro de cada unidade de quatro bases. Outras estruturas também podem ser formados, com o conjunto central de quatro bases a vir quer de um único fio dobrado em torno das bases ou de vários fios paralelos diferentes, cada uma contribuindo com uma base para a estrutura central.
Além destas estruturas empilhadas, os telómeros também formam estruturas em laço grandes chamados telomere loops ou t-loops. Aqui, os cachos de DNA de cadeia ao redor em um círculo grande estabilizado por proteínas que se ligam a telómeros. No final do T-loop, o ADN de cadeia simples dos telómeros é realizada sobre uma região de ADN de cadeia dupla pela cadeia do telómero interromper o emparelhamento de bases e o ADN de dupla hélice com uma das duas cadeias. Este estrutura de cadeia tripla é chamada de laço de deslocamento ou D-loop.
 |  |
| Única filial | Vários ramos |
DNA ramificada
No DNA o desgaste ocorre quando existem regiões não complementares no final de uma outra forma de cadeia dupla complementar de DNA. No entanto, o DNA ramificado pode ocorrer se uma terceira cadeia de ADN é introduzida e contém regiões adjacentes capazes de hibridar com as regiões desgastadas de o pré-existente de cadeia dupla. Embora o exemplo mais simples de DNA ramificado envolve apenas três fitas de DNA, complexos envolvendo fios adicionais e várias filiais também são possíveis. ADN ramificado pode ser utilizado na nanotecnologia para construir formas geométricas, consulte a secção sobre utilizações de tecnologia abaixo.
Vibração
DNA pode realizar baixa frequência de movimento coletivo como observado pelo Espectroscopia Raman e analisados com um modelo de quasi-continuum.
As modificações químicas e embalagens DNA alterado
 | 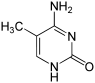 | 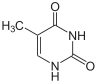 |
| citosina | 5-metilcitosina | timina |
Modificações de bases e embalagens DNA
A expressão de genes é influenciada pelo modo como o ADN é empacotado em cromossomas, em uma estrutura chamada cromatina. Modificações de bases podem ser envolvidos numa embalagem, com regiões que têm fraca ou nenhuma expressão de genes que contêm geralmente níveis elevados de metilação de bases de citosina. Embalagem do ADN e a sua influência sobre a expressão do gene pode ocorrer por meio de modificações covalentes do núcleo da proteína histona em torno do qual o DNA é envolto na estrutura da cromatina ou então por remodelação levada a cabo por cromatina remodelação complexos (ver Cromatina remodelação). Existe, ainda, crosstalk entre a metilação do DNA e modificação da histona, para que eles possam afetar coordenadamente cromatina ea expressão gênica.
Para um exemplo, metilação da citosina, produz 5-metilcitosina, que é importante para Inativação do cromossomo X. O nível médio de metilação varia entre organismos - o verme Caenorhabditis elegans carece de metilação da citosina, enquanto os vertebrados têm níveis mais elevados, com um máximo de 1% do seu DNA contendo 5-metilcitosina. Apesar da importância da 5-metilcitosina, que pode desaminar para deixar uma base de timina, assim citosinas metiladas são particularmente propensas a mutações. Outras modificações de bases incluem metilação da adenina em bactérias, a presença de 5-hidroximetilcitosina no cérebro , ea a glicosilação de uracilo para produzir o "J-base" em cinetoplastídeos.
Dano
O ADN pode ser danificado por muitos tipos de mutagénicas, que mudam a sequência de DNA. Mutagénicos incluem agentes oxidantes, agentes e também de alta energia alquilantes radiação eletromagnética , tais como ultravioleta e luz Raios-X. O tipo de dano de ADN produzido depende do tipo de agente mutagénico. Por exemplo, a luz UV pode danificar o ADN através da produção dímeros de timina, que são ligações cruzadas entre bases de pirimidina. Por outro lado, tais como oxidantes radicais livres ou peróxido de hidrogénio produzem múltiplos tipos de danos, incluindo modificações de bases, particularmente de guanosina, e quebras de cadeia dupla. Uma célula humana típica contém cerca de 150.000 bases que sofreram danos oxidativos. Destas lesões oxidativas, os mais perigosos são quebras de cadeia dupla, pois estas são difíceis de reparar e podem produzir mutações pontuais, inserções e deleções da sequência de ADN, bem como translocações cromossómicas. Estas mutações podem causar câncer . Devido às limitações inerentes aos mecanismos de reparo do DNA, se os seres humanos viveram tempo suficiente, todos iriam eventualmente desenvolver câncer. Danos de ADN que são de ocorrência natural, devido aos processos celulares normais que produzem espécies reactivas de oxigénio, as actividades hidrolíticas de água celular, etc, também ocorrem com frequência. Embora a maioria destes danos são reparados, em qualquer célula algum dano ao DNA pode permanecer, apesar da mobilização de processos de reparação. Estes danos de DNA restantes acumulam com a idade em tecidos pós-mitóticas de mamíferos. Esta acumulação parece ser uma importante causa subjacente de envelhecimento.
Muitos agentes mutagénicos encaixar-se no espaço entre dois pares de bases adjacentes, isto é chamado intercalação. A maioria dos intercaladores são moléculas aromáticas e planas; exemplos incluem brometo de etídio, acridinas, daunomicina, e doxorrubicina. Para um intercalador para caber entre pares de bases, as bases devem separar, distorcendo as fitas de DNA, desenrolando da dupla hélice. Isto inibe tanto a transcrição e a replicação do ADN, causando toxicidade e mutações. Como resultado, intercaladores de ADN podem ser cancerígenos, e no caso da talidomida, um teratogênico. Outros, como o benzo [a] pireno diol e epóxido adutos de DNA formulário aflatoxina que induzem a erros na replicação. No entanto, devido à sua capacidade para inibir a transcrição do ADN e a replicação, outras toxinas semelhantes são também utilizados em quimioterapia para inibir rápido crescimento do cancro células.
As funções biológicas
ADN geralmente ocorre como linear cromossomos em eucariotas , e cromossomas circulares em procariotas. O conjunto de cromossomos de uma célula torna-se sua do genoma; o genoma humano tem cerca de 3 bilhões de pares de bases de DNA dispostos em 46 cromossomas. A informação transportada pelo DNA é realizada no seqüência de pedaços de DNA chamados genes. A transmissão de informação genética nos genes é conseguido através de emparelhamento de bases complementares. Por exemplo, na transcrição, quando uma célula usa a informação em um gene, a sequência de ADN é copiada para uma sequência de ARN complementar através da atracção entre o ADN e os nucleótidos de RNA correctas. Normalmente, esta cópia de RNA é então usado para fazer uma correspondência sequência de proteína num processo denominado tradução, o que depende da mesma interacção entre os nucleótidos de RNA. De forma alternativa, uma célula pode simplesmente copiar a sua informação genética em um processo chamado de replicação de ADN. Os detalhes destas funções são abordados em outros artigos; aqui vamos nos concentrar sobre as interacções entre DNA e outras moléculas que medeiam a função do genoma.
Os genes e genomas
O DNA genômico é rigidamente e ordenada embalado no processo chamado A condensação de ADN para ajustar os pequenos volumes disponíveis da célula. Em eucariotas, o ADN está localizado na núcleo da célula, bem como pequenas quantidades em mitocôndrias e cloroplastos. Em procariotas, o ADN é mantido dentro de um corpo de forma irregular no citoplasma chamada nucleoid. A informação genética em um genoma é mantido dentro de genes, eo conjunto completo desta informação num organismo é chamado de genótipo. Um gene é uma unidade de hereditariedade e é uma região de ADN que influencia uma característica em particular num organismo. Os genes contêm um quadro de leitura aberto que pode ser transcrito, bem como sequências regulatórias, tais como promotores e potenciadores, que controlam a transcrição da grelha de leitura aberta.
Em muitas espécies , apenas uma pequena fracção da sequência total da genoma codifica a proteína. Por exemplo, apenas cerca de 1,5% do genoma humano consiste de codificação da proteína- exões, com mais de 50% de ADN humano que consiste em não codificante sequências repetitivas. As razões para a presença de tanto DNA não codificante em genomas eucarióticos e as diferenças extraordinárias em tamanho do genoma, ou Valor-C, entre espécies representam um enigma de longa data conhecido como o " Valor-C enigma ". No entanto, algumas sequências de DNA que não codificam proteínas podem ainda codificar funcional não codificante moléculas de ARN, as quais estão envolvidas na regulação da expressão do gene.
Algumas sequências de DNA não-codificante papel estrutural nos cromossomas. Telômeros e centrómeros contêm tipicamente poucos genes, mas são importantes para a função e estabilidade dos cromossomas. Uma forma abundante de DNA não-codificante em humanos são pseudogenes, que são cópias de genes que foram desabilitados por mutação. Estas sequências são normalmente apenas moleculares fósseis , embora possam ocasionalmente servir como material genético em bruto para a criação de novos genes com o processo de duplicação de genes e divergência.
A transcrição e tradução
Um gene é uma sequência de ADN que contém informação genética e pode influenciar o fenótipo de um organismo. Dentro de um gene, a sequência de bases ao longo de uma cadeia de ADN define um sequência de ARN mensageiro, que, em seguida, define uma ou mais sequências de proteínas. A relação entre as sequências de nucleótidos dos genes e os aminoácidos de sequências de proteínas é determinado pelas regras de tradução, conhecidos colectivamente como o código genético . O código genético consiste de três letras 'palavras' chamados códons formadas por uma sequência de três nucleótidos (por exemplo ACT, CAG, TTT).
Na transcrição, os codões de um gene são copiados para o RNA mensageiro por Polimerase de ARN. Esta cópia de RNA é então descodificada por um ribossoma que lê a seqüência de RNA por emparelhamento de bases do RNA mensageiro para ARN de transferência, que carrega aminoácidos. Uma vez que existem quatro bases em combinações de 3 letras, há 64 codões possíveis ( 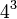 combinações). Estas codificam a vinte aminoácidos padrão, dando a maioria dos aminoácidos mais do que um codão possível. Há também três 'stop' ou códons 'nonsense' significando o fim da região de codificação; estes são o TAA, TGA e TAG códons.
combinações). Estas codificam a vinte aminoácidos padrão, dando a maioria dos aminoácidos mais do que um codão possível. Há também três 'stop' ou códons 'nonsense' significando o fim da região de codificação; estes são o TAA, TGA e TAG códons.
Réplica
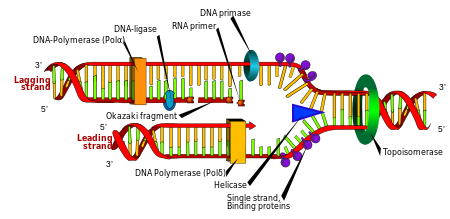
A divisão celular é essencial para que um organismo a crescer, mas, quando uma célula se divide, deve replicar o ADN no seu genoma de modo a que as duas células filhas têm a mesma informação genética como o seu progenitor. A estrutura de cadeia dupla de DNA fornece um mecanismo simples para A replicação do ADN. Aqui, as duas cadeias são separadas e, em seguida, cada um dos filamentos de seqüência de DNA complementar é recriado por um enzima chamada DNA polimerase. Esta enzima faz com que a cadeia complementar por encontrar a base correcta através de emparelhamento de bases complementares, e ligando-o para a cadeia original. Como ADN-polimerases só pode estender uma cadeia de ADN numa direcção 5 'para 3', diferentes mecanismos são usados para copiar a cadeia antiparalela da dupla hélice. Desta forma, a base sobre a antiga dita que base vai vertente vai aparecer na nova cadeia ea célula acaba com uma cópia perfeita do seu DNA.
Interacções com proteínas
Todas as funções de ADN depender de interacções com proteínas. Estes interacções entre proteínas pode ser não específica, ou a proteína pode ligar-se especificamente a uma sequência única de ADN. As enzimas também podem ligar-se ao ADN e destes, as polimerases que copiam as sequências de ADN em replicação e transcrição de ADN são particularmente importantes.
Proteínas de ligação de ADN
 |
Proteínas estruturais que se ligam ao DNA são exemplos bem compreendidos de interacções ADN-proteína não-específicos. Dentro de cromossomas, o DNA é realizado em complexos com proteínas estruturais. Estas proteínas organizar o ADN numa estrutura compacta chamada cromatina. Em eucariontes esta estrutura envolve a ligação do ADN a um complexo de pequenas proteínas básicas chamada histonas, enquanto em procariotas vários tipos de proteínas estão envolvidas. As histonas formam um complexo em forma de disco, o nucleossoma, que contém duas voltas completas de DNA de cadeia dupla enrolado à sua volta. Estas interacções não específicas são formados através de resíduos básicos na tomada de histonas ligações iônicas para a espinha dorsal ácido açúcar-fosfato do DNA, e por isso são largamente independentes da sequência de bases. As modificações químicas destes resíduos de aminoácidos básicos incluem metilação, e fosforilação acetilação. Estas modificações químicas alteram a força da interacção entre o ADN e as histonas, tornando o DNA mais ou menos acessível aos factores de transcrição e alterando a taxa de transcrição. Outras proteínas de ligação de ADN não específicos em cromatina incluem as proteínas do grupo de alta mobilidade, que se ligam ao ADN dobrado ou distorcido. Estas proteínas são importantes pois dobram conjuntos de nucleossomas e organizando-os em estruturas maiores que compõem cromossomos.
Um grupo distinto de proteínas de ligação ao ADN são as proteínas de ligação de ADN que se ligam especificamente a ADN em cadeia simples. Nos seres humanos, a replicação Uma proteína é o membro mais entendido desta família e é usado em processos onde a dupla hélice se encontre separado, incluindo a replicação do ADN, a recombinação e a reparação do ADN. Estas proteínas parecem estabilizar o ADN de cadeia simples e protegê-lo contra formando stem-loops ou de ser degradado por nucleases.

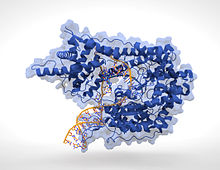
Em contraste, outras proteínas evoluíram para se ligar a sequências específicas de ADN. O mais intensivamente estudados destes são os vários factores de transcrição, que são proteínas que regulam a transcrição. Cada factor de transcrição se liga a um conjunto específico de sequências de DNA e activa ou inibe a transcrição de genes que têm essas sequências perto de seus promotores. Os fatores de transcrição fazer isso de duas maneiras. Em primeiro lugar, eles podem ligar-se a ARN-polimerase responsável pela transcrição, quer directamente ou através de outras proteínas do mediador; este localiza o polimerase no promotor e permite que ele comece a transcrição. Em alternativa, os factores de transcrição podem ligar enzimas que modificam as histonas no promotor. Isto altera a acessibilidade do modelo de ADN para a polimerase.
Como estes alvos de ADN pode ocorrer ao longo de um genoma de um organismo, alterações na actividade de um tipo de factor de transcrição pode afectar milhares de genes. Consequentemente, estas proteínas são muitas vezes os alvos dos processos de transdução de sinal que controlam respostas a mudanças ambientais ou diferenciação e desenvolvimento celular. A especificidade das interacções destes factores de transcrição 'com ADN provenientes de proteínas que fazem contactos múltiplos com os bordos das bases de DNA, permitindo a "leitura", a sequência de ADN. A maioria destas interacções-base são feitas no sulco principal, em que as bases são mais acessíveis.
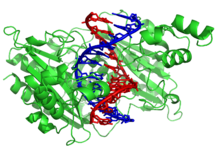
Enzimas modificadoras de DNA
As nucleases e ligases
As nucleases são enzimas que cortam as cadeias de ADN, catalisando a hidrólise das ligações fosfodiéster. As nucleases que hidrolisam nucleótidos das extremidades de cadeias de ADN são chamados exonucleases, enquanto endonucleases de corte dentro de fios. As nucleases utilizadas com mais frequência em biologia molecular são as enzimas de restrição, que cortam o ADN em sequências específicas. Por exemplo, a enzima EcoRV mostrado à esquerda reconhece a sequência de 6 bases 5'-GATATC-3 'e faz um corte na linha vertical. Na natureza, estas enzimas protegem as bactérias contra infecção fago por digestão do ADN do fago quando entra na célula bacteriana, agindo como parte do sistema de modificação de restrição. Na tecnologia, essas nucleases específica de sequências são usadas em clonagem molecular e DNA fingerprinting.
Enzimas chamadas DNA ligases podem reunir pedaços de ADN cortados ou quebrados. Ligases são particularmente importantes na replicação do ADN da cadeia atrasada, já que unem os segmentos curtos de ADN no garfo de replicação em uma cópia completa do molde de ADN. Eles são também utilizados na reparação do ADN e na recombinação genética.
Topoisomerases e helicases
As topoisomerases são enzimas que possuem a actividade de nuclease e ligase. Estas proteínas de alterar a quantidade de ADN superenrolado. Algumas destas enzimas funcionam cortando a hélice de DNA e permitindo que uma secção de rodar, reduzindo assim o seu nível de super-enrolamento; a enzima então veda a quebra de DNA. Outros tipos de enzimas são capazes de cortar uma hélice de DNA e, em seguida, passar a segunda cadeia de ADN através desta quebra, antes de reunir as hélices. As topoisomerases são necessárias para muitos processos que envolvem o ADN, tais como a replicação e transcrição de ADN.
As helicases são proteínas que são um tipo de motor molecular. Eles usam a energia química em trifosfatos de nucleósidos, predominantemente ATP , para romper as ligações de hidrogênio entre as bases e descontrair dupla hélice do ADN em cadeias simples. Estas enzimas são essenciais para a maioria dos processos em que as enzimas necessitam de aceder às bases do ADN.
Polimerases
As polimerases são enzimas que sintetizam cadeias de polinucleótidos a partir de nucleósido-trifosfatos. A sequência de seus produtos são cópias de cadeias-polinucleot�icas existentes que são chamados modelos . Estas enzimas funcionar por meio de adição de nucleótidos para a 3 ' do grupo hidroxilo do nucleótido anterior numa cadeia de ADN. Como uma consequência, todas as polimerases de trabalhar numa direcção 5 'para 3'. No local activo destas enzimas, os nucleósido-trifosfato de entrada de pares de bases para o molde: esta permite polimerases com precisão para sintetizar a cadeia complementar do seu molde. Polimerases são classificados de acordo com o tipo de modelo que eles usam.
Na replicação do ADN, dependente de ADN, uma ADN-polimerase faz uma cópia de uma sequência de ADN. A precisão é vital neste processo, por isso muitas destas polimerases têm uma atividade de revisão. Aqui, a polimerase reconhece erros ocasionais na reacção de síntese por a falta de emparelhamento de bases entre os nucleótidos desemparelhados. Se for detectada alguma diferença, uma 3 'para 5' exonuclease é activado e a base incorrecta removido. Na maioria dos organismos, função de polimerases de ADN em um grande complexo denominado replissoma que contém múltiplas unidades acessórias, tais como a braçadeira de ADN ou helicases.
ADN-polimerases dependentes de ARN são uma classe especializada de polimerases que copiam a sequência de uma cadeia de ARN em ADN. Eles incluem transcriptase reversa, que é um vírus enzima envolvida na infecção de células por retrovírus, e da telomerase, que é necessário para a replicação dos telómeros . A telomerase é uma polimerase inusual, porque contém o seu próprio molde de ARN como parte de sua estrutura.
A transcrição é realizada por uma ADN-dependente da polimerase de ARN que copia a sequência de uma cadeia de DNA em RNA. Para iniciar a transcrição de um gene, a ARN-polimerase se liga a uma sequência de ADN denominada promotor, e separa as cadeias de ADN. É então cópias da sequência do gene num transcrito de ARN mensageiro até atingir uma região de ADN chamado o terminador, onde se detém e se separa do ADN. Tal como acontece com as polimerases de ADN humano dependente de DNA, RNA polimerase II, a enzima que transcreve a maioria dos genes no genoma humano, opera como parte de um grande complexo de proteína com múltiplas subunidades reguladoras e acessórios.
A recombinação genética
 |
 |
Uma hélice de ADN normalmente não interage com outros segmentos de DNA, e em células humanas os diferentes cromossomas ocupam áreas separadas no núcleo chamado de "territórios cromossómicos". Esta separação física dos diferentes cromossomas é importante para a capacidade do ADN para funcionar como um repositório estável para informação, como uma das poucas vezes é cromossomas interagem durante cruzamento cromossómico quando eles se recombinam. Cruzamento cromossómico é quando duas hélices de ADN se rompem, sofrem intercâmbio e se unem novamente.
A recombinação permite cromossomos para a troca de informação genética e produz novas combinações de genes, o que aumenta a eficiência da seleção natural e pode ser importante na rápida evolução de novas proteínas. A recombinação genética pode também ser envolvido na reparação do ADN, particularmente na resposta da célula a quebras de cadeia dupla.
A forma mais comum de cruzamento cromossómico é a recombinação homóloga, em que as duas partes envolvido cromossomas sequências muito semelhantes. A recombinação não-homóloga pode ser prejudicial para as células, uma vez que pode produzir translocações cromossómicas e anormalidades genéticas. A reacção de recombinação é catalisada por enzimas conhecidas como recombinases, tais como RAD51. O primeiro passo no processo de recombinação é uma quebra de cadeia dupla causado por qualquer um de endonuclease ou danos no ADN. Uma série de passos catalisados em parte pela recombinase, em seguida, leva à união das duas hélices por, pelo menos, uma junção Holliday, em que um segmento de um único cordão em cada hélice é fundido com a cadeia complementar na outra hélice. A junção Holliday é uma estrutura de união tetraédrica que pode ser movido ao longo do par de cromossomas, a troca de um fio para outro. A reacção de recombinação é então interrompida por clivagem da junção e re-ligação do ADN libertado.
Evolução
DNA contém a informação genética que permite que todos os modernos seres vivos funcionar, crescer e se reproduzir. No entanto, não está claro quanto tempo no 4 bilhões de anos da história de DNA vida executou esta função, como tem sido proposto que as primeiras formas de vida poderiam ter utilizado ARN como material genético. RNA pode ter agido como a parte central do início metabolismo celular, uma vez que pode transmitir informação genética e realizar catálise como parte de ribozimas. Este antigo mundo de ARN onde o ácido nucleico teria sido usada tanto para catálise e genética podem ter influenciado a evolução do código genético atual baseada em quatro bases de nucleotídeos. Isto poderia ocorrer, uma vez que o número de bases diferentes em tal organismo é um trade-off entre um pequeno número de bases de precisão cada vez maior de replicação e um grande número de bases de aumentar a eficiência catalítica das ribozimas.
No entanto, não existe qualquer evidência directa de sistemas genéticos antigos, como a recuperação de ADN a partir de a maioria dos fósseis é impossível. Isto é porque o DNA sobrevive no ambiente por menos de um milhão de anos e lentamente degrada-se em pequenos fragmentos em solução. Pedidos de indemnização por DNA mais velho foram feitas, mais notavelmente um relatório do isolamento de uma bactéria viável a partir de um cristal de sal 250 milhões anos de idade, mas essas alegações são controversos.
Em 8 de agosto de 2011, um relatório, com base naNASAestudos commeteoritos encontrados naTerra, foi publicado sugerindo blocos de construção de DNA ( adenina, guanina e afinsmoléculas orgânicas) podem ter sido formados em extraterrestre espaço sideral.
Usa em tecnologia
Engenharia genética
Foram desenvolvidos métodos para purificar DNA a partir de organismos, tais como a extracção com fenol-clorofórmio, e manipulá-lo em laboratório, tais como digestões de restrição e a reacção em cadeia da polimerase . Moderna biologia e bioquímica fazem uso intensivo destas técnicas em tecnologia de ADN recombinante. ADN recombinante é uma sequência de ADN artificial que foi montado a partir de outras sequências de ADN. Eles podem ser transformados em organismos sob a forma de plasmídeos ou no formato apropriado, usando um vector viral. O organismos geneticamente modificados produzidos podem ser utilizados para produzir produtos, tais como recombinantes de proteínas , utilizados na pesquisa médica, ou ser cultivadas em agricultura .
Forensics
Os cientistas forenses pode usar DNA em sangue , sêmen, pele, saliva ou cabelo encontrado em uma cena de crime para identificar um DNA correspondente de um indivíduo, como um agressor. Este processo é denominado formalmente perfis de DNA, mas pode também ser chamado " impressão digital genética ". Em perfis de ADN, os comprimentos das secções variáveis de DNA repetitivo, como repetições curtas em tandem e minissatélites, são comparados entre as pessoas. Este método é normalmente uma técnica muito fiável para identificar um ADN correspondente. No entanto, a identificação pode ser complicado se a cena está contaminada com ADN de pessoas diferentes. Perfis de ADN foi desenvolvida em 1984 pelo geneticista britânico Sir Alec Jeffreys, e usado pela primeira vez em medicina forense para condenar Colin Pitchfork no 1.988 assassinatos caso Enderby.
O desenvolvimento da ciência forense, ea capacidade de obter agora correspondência genética em amostras de sangue minutos, pele, saliva ou cabelo levou a uma re-análise de um número de casos. Evidência agora pode ser descoberto que não foi cientificamente possível no momento do exame inicial. Combinado com a remoção da lei dupla penalização em alguns lugares, isso pode permitir que os processos ser reaberto em que estudos anteriores não conseguiram produzir provas suficientes para convencer um júri. Pessoas acusadas de crimes graves podem ser obrigados a fornecer uma amostra de DNA para fins de correspondência. A defesa mais óbvio para partidas de DNA obtidos forense é a alegação de que a contaminação cruzada de provas teve lugar. Isso resultou em procedimentos de manipulação rigorosos meticulosos com novos casos de criminalidade grave. Perfis de DNA também é utilizado para identificar vítimas de incidentes de desastre em massa. Bem como identificar positivamente órgãos ou partes do corpo em acidentes graves, perfis de DNA está sendo usado com sucesso para identificar vítimas individuais em túmulos de guerra em massa - correspondência aos membros da família.
Bioinformática
Bioinformática envolve a manipulação, busca e mineração de dados de dados biológicos, e isso inclui dados de sequência de DNA. O desenvolvimento das técnicas para armazenar e procurar sequências de ADN gerou avanços amplamente aplicados em ciência da computação , especialmente corda busca algoritmos, aprendizado de máquina e teoria de banco de dados. Seqüência de pesquisa ou algoritmos, que procuram a ocorrência de uma sequência de letras dentro de uma sequência de letras maior harmonização, foram desenvolvidos para procurar sequências específicas de nucleótidos. A sequência de ADN pode ser alinhadas com outras sequências de ADN para identificar sequências homólogas e localizar específicas de mutações que os tornam distintos. Estas técnicas, especialmente o alinhamento de sequências múltiplas , são utilizados para estudar as relações filogenéticas e função da proteína. Conjuntos de dados que representam a pena 'genomas inteiros de sequências de ADN, como os produzidos pelo Projeto Genoma Humano, são difíceis de usar sem as anotações que identificam os locais de genes e elementos reguladores em cada cromossoma. As regiões de ADN que possuem os padrões característicos associados com genes de proteínas ou que codifica a ARN podem ser identificados por algoritmos de localização de genes, que permitem que os investigadores para prever a presença de determinados produtos de genes e as suas possíveis funções num organismo, mesmo antes de ter sido isolado experimentalmente. Genomas inteiros também podem ser comparados, o que pode lançar luz sobre a história evolutiva de particular organismo e permitir o controlo dos acontecimentos evolutivos complexos.
Nanotecnologia de DNA

Nanotecnologia ADN utiliza as únicas propriedades de reconhecimento molecular de ADN e outros ácidos nucleicos para criar complexos de ADN ramificadas auto-montagem com propriedades úteis. ADN é assim utilizada como um material estrutural, em vez de como um portador de informação biológica. Isto levou à criação de reticulados periódicos bidimensionais (tanto à base de telha, bem como utilizando o " origami de DNA "método), bem como estruturas tridimensionais nas formas de poliedros . Nanomechanical e dispositivos de auto-montagem algorítmica também têm foi demonstrado, e estas estruturas de DNA foram usadas para o arranjo de molde de outras moléculas, tais como as nanopartículas de ouro e proteínas estreptavidina.
História e antropologia
Como o DNA armazena mutações ao longo do tempo, que são então herdadas, que contém informações históricas, e, comparando seqüências de DNA, os geneticistas podem inferir a história evolutiva dos organismos, o seu filogenia. O campo da filogenia é uma ferramenta poderosa na biologia evolutiva . Se as sequências de ADN dentro de uma espécie são comparados, os geneticistas de populações podem conhecer a história de populações particulares. Isso pode ser usado em estudos que vão desde a ecologia genética para a antropologia ; Por exemplo, evidências de DNA está sendo usada para tentar identificar as dez tribos perdidas de Israel.
DNA também tem sido usado para olhar para as relações familiares modernos, tais como o estabelecimento de relações de parentesco entre os descendentes de Sally Hemings e Thomas Jefferson . Este uso está intimamente relacionado com o uso de DNA em investigações criminais detalhados acima. De fato, algumas investigações criminais foram resolvidos quando o DNA de cenas de crime tem acompanhado parentes do indivíduo culpado.
Armazenamento de informação
Em um artigo publicado na revista Nature em janeiro de 2013, cientistas dos Bioinformatics Institute e europeus Agilent Technologies propôs um mecanismo para usar a capacidade do DNA para codificar informações como um meio de armazenamento de dados digital. O grupo foi capaz de codificar 739 kilobytes de dados em código de DNA, sintetizar o DNA real, então sequenciar o DNA e decodificar as informações de volta à sua forma original, com uma precisão de 100% reportado. A informação codificada consistiu de arquivos de texto e arquivos de áudio. Um experimento anterior foi publicado em agosto de 2012. O estudo foi realizado por pesquisadores da Universidade de Harvard, onde o texto de um livro de 54.000 palavras foi codificada no DNA.
História da pesquisa de DNA

O DNA foi isolado pela primeira vez pelo suíço médico Friedrich Miescher que, em 1869, descobriu uma substância microscópico no pus de ataduras cirúrgicas descartados. Como ele residiu no núcleo das células, ele chamou de "nucleína". Em 1878, Albrecht Kossel isolado o componente não proteico de "nucleína", o ácido nucleico, e mais tarde isolado seus cinco primárias nucleobases. Em 1919, Phoebus Levene identificou a unidade de base, açúcar e fosfato de nucleótidos. Levene sugeriu que o DNA consistia em uma série de unidades de nucleotídeos ligados entre si através dos grupos fosfato. No entanto, pensou Levene cadeia curta foi repetido e as bases numa ordem fixa. Em 1937 William Astbury produziu os primeiros padrões de difracção de raios X que mostraram que o DNA tinha uma estrutura regular.
Em 1927, Nikolai Koltsov propôs que traços herdados seriam herdadas por meio de uma "molécula hereditária gigante" composta de "duas vertentes espelho que iria replicar de forma semi-conservador utilizando cada fio como um modelo". Em 1928, Frederick Griffith descobriu que traços da "suave" forma de pneumococo poderão ser transferidos para a forma "grosseira" da mesma bactéria através da mistura de mortos bactérias "liso" com a forma viva "áspero". Este sistema proporcionou a primeira sugestão clara de que o DNA transporta informação genética-o Avery-MacLeod-McCarty experimento, quando Oswald Avery, juntamente com colegas de trabalho Colin MacLeod e McCarty Maclyn, ADN identificado como o princípio de transformação em função do DNA em 1943. hereditariedade foi confirmado em 1952, quando Alfred Hershey e Martha perseguição no experimento Hershey-chase mostrou que o DNA é o material genético do fago T2.
Em 1953,James D. WatsoneFrancis Cricksugeriu que hoje é aceita como o primeiro modelo de dupla hélice correta daestrutura do DNA no jornal Nature.A dupla hélice, modelo molecular do DNA foi então baseada em uma únicaimagem de difração de raios-X ( rotulado como "Foto 51 ") feita pelo Rosalind Franklin e Raymond Gosling maio 1952, bem como as informações que as bases de ADN são emparelhados - também obtidas através de comunicações privadas deErwin Chargaff nos anos anteriores.regras de Chargaff desempenhou um papel muito importante no estabelecimento de casal configurações hélice para B-DNA, bem como um ADN-.
A evidência experimental apoiando a Watson e Crick modelo foi publicado em uma série de cinco artigos na mesma edição da Nature . Destes, papel de Franklin e Gosling foi a primeira publicação de seus próprios dados de difração de raios-X e método de análise original que apoiou parcialmente o modelo de Watson e Crick; esta questão também continha um artigo sobre a estrutura do DNA por Maurice Wilkins e dois de seus colegas, cuja análise e in vivo padrões B-DNA de raios-X também apoiou a presença in vivo das configurações de dupla hélice de DNA como proposto por Crick e Watson para seu modelo molecular de dupla hélice do DNA nas duas páginas anteriores de Natureza . Em 1962, após a morte de Franklin, Watson, Crick e Wilkins receberam conjuntamente o Prêmio Nobel de Fisiologia ou Medicina. Prêmios Nobel foram concedidos apenas para os destinatários vivendo no momento. Um debate continua sobre quem deveria receber crédito pela descoberta.
Em uma apresentação influente em 1957, Crick estabeleceu o dogma central da biologia molecular, que predisse a relação entre o DNA, RNA e proteínas, e articulou a "hipótese adaptador". A confirmação final do mecanismo de replicação que foi implicado pela estrutura de dupla hélice seguido em 1958 por meio da experiência de Meselson-Stahl. Mais trabalho por Crick e colaboradores mostraram que o código genético foi baseado em tripletos não sobrepostos de bases, chamados codões, permitindo Har Gobind Khorana, Robert W. Holley e Marshall Warren Nirenberg para decifrar o código genético. Estes resultados representam o nascimento de Biologia molecular.

